Bioinformatica
-
Biologia
-
Classi: 5° anno
-
-
-
Laboratorio virtuale
-
Percorso didattico
-
2 h
-
Min. 3 persone
-
Nessuna
Riassunto / Abstract
Questa attività trae spunto dal progetto Openlab (http://www.openlab.unifi.it/mdswitch.html) svolto al polo ST a Sesto Fiorentino con il Prof. Marco Fondi. Si tratta di usare la banca dati open source NCBI e specifici programmi computazionali per l’analisi di sequenze di DNA. Le metodiche che sfruttando l’informatica permettono di analizzare e comparare velocemente tratti di DNA di varie specie, catalogare tratti codificanti e non, individuare i tratti di DNA che possono codificare una proteina partendo dalla sua sequenza. Attraverso questa esperienza, i ragazzi possono prendere contatto con alcune metodiche di "genetica inversa", possono capire cosa significa analizzare i processi evolutivi attraverso le sequenze del DNA e comprendere il significato di "speciazione".
Scheda sintetica delle attività
Useremo Basic Local Alignment Search Tool BLAST e NCBI http://www.ncbi.nlm.nih.gov/
Perché comparare le sequenze usando l’informatica?
- Per individuare quali altri organismi possiedono il gene sotto studio (query, seed) (es. produzione antibiotici, target per farmaci)
- Per una preliminare assegnazione di una struttura proteica ad una sequenza genica (genetica inversa)
- Per una preliminare attribuzione funzionale (hypothetical protein, putative function)
Come lavora BLAST? Si compone di tre fasi:
- Scomposizione della query
- Ricerca di un appaiamento
- Estensione dell’allineamento
All'interno dell'esperienza, faremo anche un confronto tra cariotipi di diversi organismi.
Si possono confrontare il numero e struttura dei cromosomi di organismi diversi nel SITO "https://www.ensembl.org/index.html"
Si possono confrontare il numero e struttura dei cromosomi di organismi diversi nel SITO "https://www.ensembl.org/index.html"
- Attività 1. Si parte da una sequenza proteica e si vanno a cercare gli organismi che presentano sequenze simili. Si discute degli score e dei valori percentuali di similitudine trovati.
- Attività 2. Si parte invece da una sequenza nucleotidica e analogamente si cercano paragoni con sequenze di altre specie
- Attività 3. Si mette a paragone direttamente un tratto di cromosoma tra specie diverse.
In aggiunta a questo, si possono lasciare i ragazzi liberi di discutere circa le affinità riscontrate e soprattutto dei vantaggi che offre oggi l'informatica nell'ambito della ricerca.
Risorse necessarie
- Computer;
- collegamento a rete internet.
Prerequisiti necessari
- Conoscenze sulla sequenza amminoacidica e nucleotidica;
- conoscenza del codice genetico;
- conoscenze sulla sintesi delle proteine.
Obiettivi di apprendimento
- Capacità di indagine sulle banche dati;
- capacità di sfruttare la velocità del computer nell’analisi di sequenze;
- capacità di trarre conclusioni efficaci dalle osservazione di confronti tra sequenze.
Dotazioni di sicurezza
Nessuna
Svolgimento
Attività 1
Si accede al sito http://www.ncbi.nlm.nih.gov/ si apre il menù a tendina e si seleziona Protein e nel settore di ricerca "HIV-1"[organism] NOT srcdb_refseq[properties]. Si apre la banca dati dove ci sono varie notizie; a noi interessa la SEQUENZA da FASTA che andiamo a copiare su un file di testo ".TXT" oppure si può salvare dalla apposita finestra a destra. A questo punto torniamo indietro e apriamo BLAST e incolliamo la sequenza amminoacidica in blastp. Il primo form in alto serve ad incollare la sequenza che vogliamo analizzare. Nel secondo blocco, subito sotto al primo, possiamo scegliere il database che verrà utilizzato dall'algoritmo ed eventualmente, specificare l'organismo su cui vogliamo che si concentri la ricerca. Infine, nel terzo blocco possiamo scegliere l'algoritmo che verrà usato. Una volta eseguite queste operazioni, possiamo lanciare il programma cliccando sul tasto BLAST. Una volta conclusa la fase di ricerca, si apre il rapporto che evidenzia il resoconto di compatibilità (figura 1).

Elenco dei comandi presenti in BLAST:
blastp: Protein sequence (PS) query versus PS database
blastn: Nucleic acid sequence (NS) query versus NS database
blastx: NS query translated in all six reading frames versus PS database
tblastn: PS query versus NS database dynamically translated in all six reading frames
tblastx: Translated NS query versus translated NS database - computationally intensive
Attività 2
Dal menù a tendina iniziale, selezioniamo questa volta Nucleotide e nel settore di ricerca 126686248. Questa è una sequenza GSS genome survey sequences. Facendo click sul link attivo GSS si apre la descrizione del gene e varie altre notizie. A questo punto copiamo la sequenza o salviamo il file in formato FASTA.
Apriamo di nuovo BLAST e nella sezione blastn copiamo la sequenza volendola paragonare con le sequenze di DNA di topo; non viene restituito nessun risultato. Proviamo allora a eseguire una nuova ricerca inserendo altre specie. Compare uno score positivo con una certa percentuale di similitudine con un plasmodio e con un batterio causa di polmonite (figura 2).
Apriamo di nuovo BLAST e nella sezione blastn copiamo la sequenza volendola paragonare con le sequenze di DNA di topo; non viene restituito nessun risultato. Proviamo allora a eseguire una nuova ricerca inserendo altre specie. Compare uno score positivo con una certa percentuale di similitudine con un plasmodio e con un batterio causa di polmonite (figura 2).
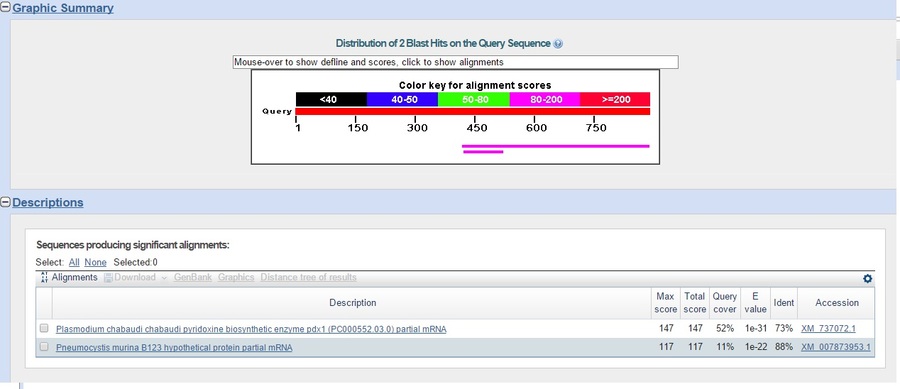
Attività 3
Cambiamo ora sito e vediamo un altro tipo di attività bioinformatica. Dalla Home page http://www.ensembl.org/index.html appare una schermata con tutte le specie presenti nel database. Possiamo scegliere Human e successivamente
cliccare sull’icona "view CARYOTYPE". Si aprirà una pagina in cui saranno presenti tutti i cromosomi (in forma aploide) dell'uomo. A questo punto si può cliccare su una sezione specifica di un cromosoma (es. cromosoma 8) e si verrà rimandati ad una pagina contenente numerose informazioni del tratto cromosomico come ad esempio: i geni presenti in quel tratto, le sequenze codificanti e non e molte altre informazioni (figura 3).
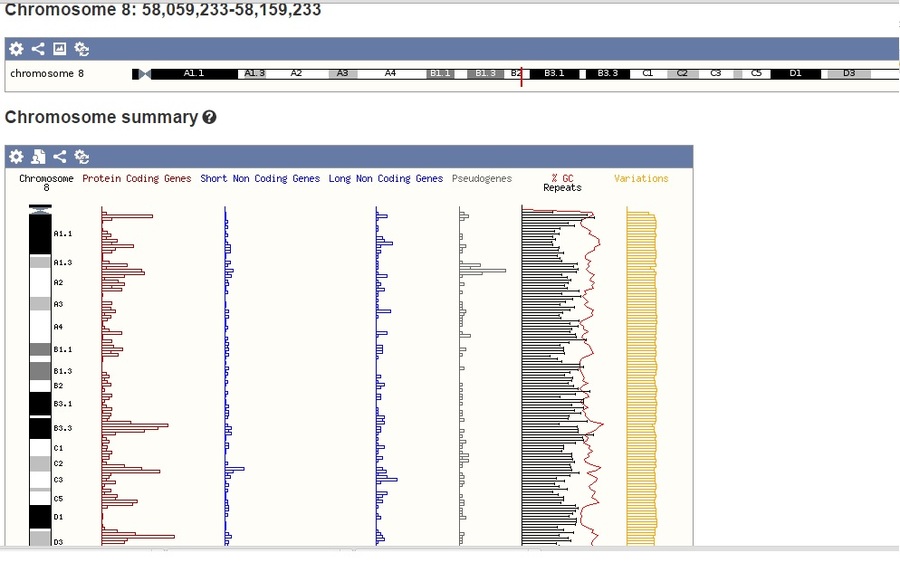
Oltre ad analizzare in maniera dettagliata la regione del cromosoma di nostro interesse, possiamo comparare cromosomi umani con quelli di altre specie. Per far questo, è sufficiente cliccare su una regione del cromosoma e scegliere dal menù a sinistra l'opzione synteny. Il programma automaticamente paragonerà la nostra regione cromosomale con le sequenze di topo restituendoci un report molto simile a quanto riportato nella figura 4. Tuttavia è possibile scegliere, dal menù a tendina presente sulla destra, anche altri organismi.

Note e storia
Questa attività vuole essere una piccola guida propedeutica all'eventuale somministrazione di quesiti d'esame di tipo informatico
Bibliografia
Autori
Burani Paola

